En los últimos años, algunas personas de ciencia han retomado el estudio del cáncer en un fenómeno fascinante y, a menudo devastador descubierto hace más de 100 años: la aneuploidía, una característica fundamental de la mayoría de los cánceres. La aneuploidía se refiere a la presencia de un número anormal de cromosomas en una célula, que promueve una inestabilidad genómica considerada uno de los sellos distintivos de muchos tipos de cáncer.
Para comprender cómo las células cancerosas sobrellevan los efectos perjudiciales de la aneuploidía, un equipo multidisciplinario de investigadores de la Universidad de Costa Rica (UCR), el Tecnológico de Costa Rica (TEC) y la Universidad Estatal a Distancia (UNED) se embarcó en un proyecto pionero. La aneuploidía permite al cáncer evolucionar hacia la malignidad pero tiene también muchos efectos negativos en las células
Se planteó entonces la hipótesis de que la compensación de la dosis de genes cruciales podría ser un mecanismo mediante el cual las células cancerosas pueden resistir los impactos negativos de la aneuploidía. Este fenómeno intrigante llevó a una investigación biocomputacional profunda, utilizando datos del panel de líneas celulares de cáncer NCI-60 y otras fuentes.
¿Qué es el panel de líneas celulares de cáncer NCI-60?
El Panel de Líneas Celulares de Cáncer NCI-60 es un conjunto de 60 líneas celulares de cáncer humano que se utiliza como una herramienta importante en la investigación oncológica y en el desarrollo de fármacos. Estas líneas celulares se derivan de varios tipos de cáncer, como el cáncer de mama, colon, pulmón, ovario y leucemia, entre otros para los que se cuenta con datos genómicos. El Panel NCI-60 ha sido una herramienta fundamental en la identificación y desarrollo de numerosos fármacos contra el cáncer y se encuentra disponible en la Universidad de Costa Rica
El objetivo central de este proyecto fue identificar a nivel computacional las redes de regulación de miARNs, lncRNAs y factores de transcripciónimplicados en la compensación de dosis génica en el contexto del cáncer aneuploide. Además, se buscó evaluar el potencial de estas redes como blancos terapéuticos mediante su inhibición en modelos experimentales del panel NCI60.
¿Qué son miARNs?
Son pequeñas moléculas de ARN (ácido ribonucleico) involucrados en la regulación de la actividad de los genes. La función principal de los miARNs es inhibir la expresión de ciertos genes al unirse a las moléculas de ARN mensajero (ARNm). Cuando un miARN se empareja con un ARNm específico, puede evitar que se traduzca en proteínas o acelerar su degradación.Los miARNs desempeñan un papel fundamental en una variedad de procesos biológicos, incluyendo el desarrollo embrionario, la diferenciación celular, la proliferación celular, la apoptosis (muerte celular programada) y la respuesta inmune.
¿Qué son IncARNs?
Son una categoría de ARN (ácido ribonucleico) que desempeñan roles reguladores en la célula. Son moléculas de ARN más largas que los miARNs (microARNs) y tienen una longitud superior a 200 nucleótidos. Participan en diversas actividades celulares, incluyendo la regulación de la expresión génica.
Los lncARNs han sido implicados en una amplia gama de procesos biológicos, como el desarrollo embrionario, la diferenciación celular, la regulación del ciclo celular y la respuesta inmune. Además, se ha demostrado que los lncARNs desempeñan un papel en diversas enfermedades, incluyendo el cáncer, enfermedades neurodegenerativas y trastornos metabólicos, lo que los convierte en objetivos de investigación importantes para comprender mejor su función y potencial terapéutico.
Para alcanzar estos objetivos, el equipo de investigación se enfocó en una serie de metas específicas y desarrolló un conjunto de indicadores para medir el progreso en cada una de estas áreas clave:
1. Identificación de Genes Bajo Compensación de Dosis Génica:
Para identificar genes sometidos a la compensación de dosis génica, se estableció un sistema de base de datos innovador. Este sistema no solo recopiló datos cruciales, sino que también permitió la validación de los genes identificados y proporcionó información valiosa sobre su función y regulación.
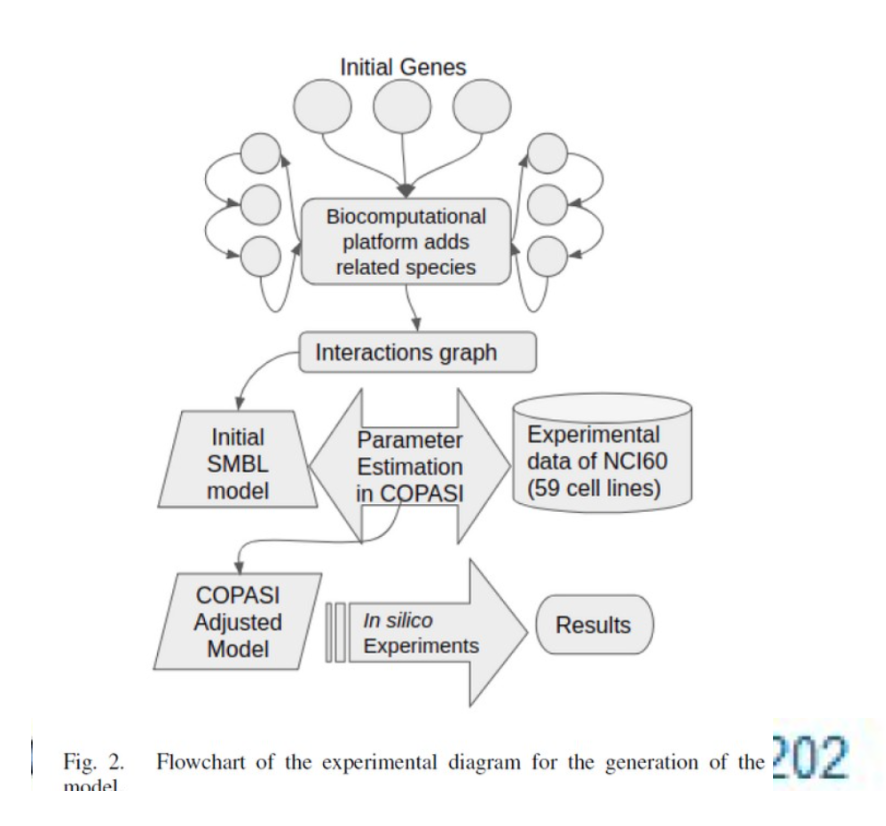
2. Desarrollo de una Plataforma Biocomputacional:
Una plataforma de modelado y simulación de sistemas biológicos complejos se implementó utilizando herramientas avanzadas como HTCondor y Cloud-COPASI. Estas herramientas permitieron la ejecución eficiente de modelos matemáticos detallados, proporcionando una comprensión profunda de las interacciones entre los miARNs, lncARNs, factores de transcripción y los genes implicados en la compensación de dosis génica.
¿Qué es Cloud Copasi?
Cloud-COPASI es una herramienta basada en web para ejecutar tareas de simulación y análisis computacionalmente intensivas en paralelo en un grupo de computación de alto rendimiento. Cloud-COPASI puede conectarse a grupos de computación existentes o proporciona una interfaz sencilla para lanzar un nuevo grupo de computación utilizando el servicio Amazon Elastic Compute Cloud (EC2).
Cloud-COPASI puede llevar a cabo diversas tareas de simulación y análisis, incluyendo análisis de sensibilidad global, simulaciones estocásticas, exploraciones de parámetros, optimizaciones y ajustes de parámetros. Cada tarea se divide automáticamente en varios trabajos más pequeños que se ejecutan en paralelo, lo que permite acelerar significativamente el tiempo de ejecución.
¿Qué es HTCondor?
HTCondor es un sistema de software que crea un entorno de cómputo de alto rendimiento (HTC). Utiliza eficazmente el poder de cómputo de máquinas conectadas en red, ya sea un solo clúster, un conjunto de clústeres en un campus, recursos en la nube o redes internacionales de cómputo. HTCondor puede aprovechar de manera efectiva los recursos compartidos con propiedad distribuida
3.Identificación de Redes de miARNs y Factores de Transcripción:
Se construyeron y analizaron redes detalladas de interacción entre miARNs y factores de transcripción. Estos modelos matemáticos no solo revelaron las complejidades de las interacciones moleculares, sino que también permitieron la identificación de nodos críticos en estas redes.
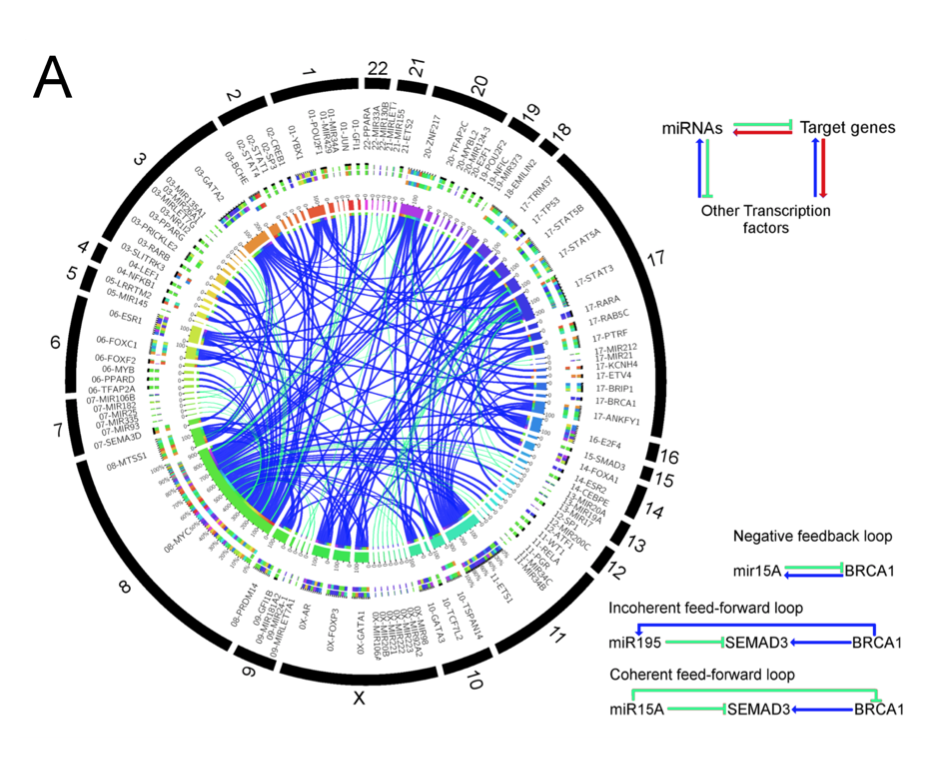
Topología de interacciones de miRNAs y factores de transcripción.
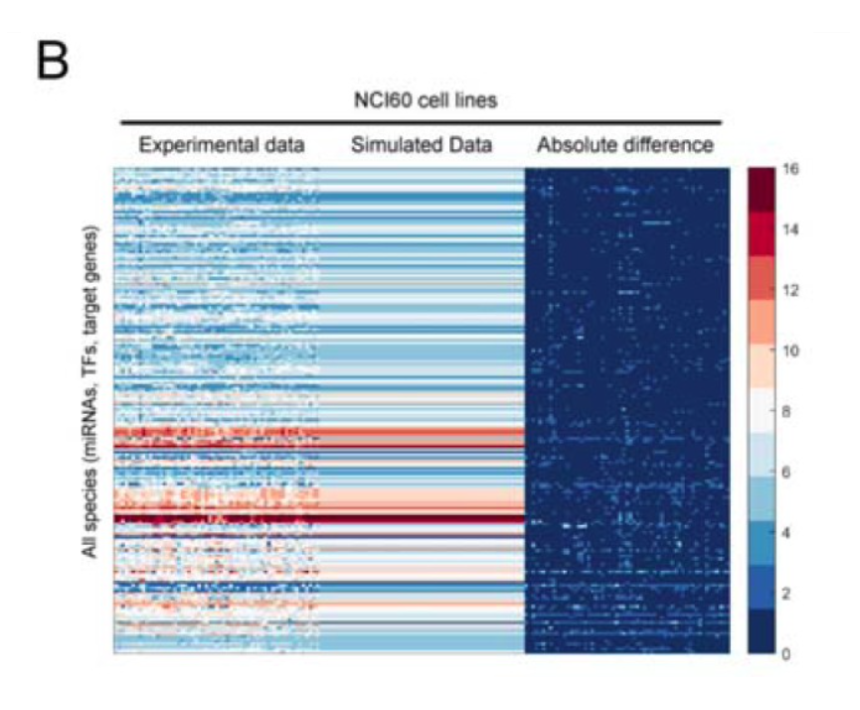
Comparación de datos experimentales y simulados por el modelo ajustado.
4.Identificación de posibles lncARNs involucrados en la Compensación de Dosis Génica:
Se desarrolló una metodología innovadora para identificar lncARNs asociados con la compensación de dosis génica. Esta metodología se basó en análisis detallados de secuencias genómicas y se mejoró continuamente para optimizar la identificación precisa de lncARNs relevantes.
5. Validación Experimental de los miARNs y lncARNs como Blancos Terapéuticos:
Se diseñaron y sintetizaron moléculas anti-miARN y anti-lncARN para evaluar sus efectos antitumorales en líneas celulares de cáncer. Estas moléculas se probaron rigurosamente, y los resultados experimentales proporcionaron una validación crucial de las predicciones computacionales.
Contribución Académica:
Esta investigación también tuvo como productos varios artículos académicos publicados, entre ellos:
- Acón MS, Siles-Canales F, Mora- Rodríguez RA. A biocomputational platform for the automated construction of large-scale mathematical models of miRNA-transcription factor networks for studies on gene dosage compensation. IEEE Xplore (ISBN 978-1- 4673-9578- 6). June 2017. doi: 10.1109/CONCAPAN.2016.7942348. Disponible en https://ieeexplore.ieee.org/document/7942348
- Oviedo-Blanco G, Acon-Chan Man-Sai, Guevara-Coto J, Mora-Rodríguez R. Analysis of Large Scale Gene Expression Data sets from TCGA Identifies Potential Candidate Genes under Dosage Compensation in Breast Cancer. Int’l Conf. Bioinformatics and Computational Biology | BIOCOMP’18 | 130-137 ISBN: 1-60132-471- . Disponible en: https://csce.ucmss.com/cr/books/2018/LFS/ CSREA2018/BIC4304.pdf
- Man-Sai Acón-Chan, Francisco Siles- Canales and Rodrigo Mora. Modeling gene dosage compensation mediated by sensor loops in large-scale mathematical models of microRNA- transcription factor networks. 2018 IEEE International Work Conference on Bioinspired Intelligence (IWOBI). ISBN 978-1-5386-7506-9. Disponible en https://ieeexplore.ieee.org/document/8464210
- Acón, ManSai, Geiß, Carsten, Torres-Calvo, Jorge, Bravo-Estupiñan, Diana, Oviedo, Guillermo, Arias-Arias, Jorge L, Rojas-Matey, Luis A, Baez, Edwin, Vásquez-Vargas, Gloriana, ,Oses-Vargas, Yendry, Guevara-Coto, José, Segura-Castillo, Andrés, Siles-Canales, Francisco, Quirós-Barrantes, Steve, Régnier-Vigouroux, Anne, Mendes, Pedro, Mora-Rodríguez, Rodrigo. MYC dosage compensation is mediated by miRNA-transcription factor interactions in aneuploid cancer. iScience 2021. https://doi.org/10.1016/j.isci.2021.103407
Este proyecto pionero ha arrojado luz sobre los misterios del cáncer aneuploide. Se ha identificado una red de interacciones complejas entre factores de transcripción y miARNs, destacando la compensación de dosis génica para genes clave como MYC y STAT3. La inhibición de miARNs compensatorios ha surgido como una estrategia terapéutica prometedora contra el cáncer aneuploide, ofreciendo nuevas perspectivas para el tratamiento de pacientes afectados por esta enfermedad compleja y desafiante.
Además, en los siguientes meses de terminado este proyecto se logró desarrollar una novedosa plataforma para la validación experimental de la compensación de dosis génica de MYC y se ha comprobado que su inhibición puede llevar a la inducción de muerte celular de forma específica en células con amplificación de este gen. Esto establece la base para una potencial aplicación terapéutica de la inhibición de la compensación de dosis génica para combatir los múltiples tipos de cáncer que presentan aneuploidías específicas.
Contacto
Rodrigo Mora Rodriguez rodrigo.morarodriguez@ucr.ac.cr
Participantes
| Rodrigo Mora Rodríguez | UCR | Facultad de Microbiología, CIET, Posgrado de Bioinformática |
| Juan Luis Crespo | TEC | Área de Ing. Mecatrónica |
| Andrés Segura | UNED | Laboratorio de Investigación e Innovación Tecnológica |
| Cindy Calderón Arce | TEC | Escuela de Matemática |
| Guillermo Oviedo Blanco | UCR | Maestría en Bioinformática |
| Man Sai Acón Chan | UCR | Maestría en Bioinformática |
Asistentes
- Gloriana Vásquez
- Andrea Rojas
- Felipe Castillo Guillén
- Arick Dickerman
- Jurguen Bermudez
